| Mais
qu'est ce qu'une puce à ADN ? (suite)
3.
La fixation de la sonde sur le support.
Il
existe deux types d'attachement des sondes sur les supports mais
plusieurs techniques en fonction du support choisi :
3a.
Fixation de l'oligonucléotide déjà synthétisé.
Les sondes synthétisées aupréalablement
sont transférées sur la puce au moyen de micropipettes,
de micropointes ou par des dispositifs de type jet d'encre pilotés
par des robots. Il existe 4 méthodes d'adressage des sondes :
 L'adressage
électrochimique. L'adressage
électrochimique.
|
La
puce est composée d'un support en silicium recouvert, à chaque
plot d'une mini-électrode en or de 50 µm. Lorsque les mini-électrodes
sont mises sous tension sélective, une électro-polymération
se produit et chaque sonde rejoint un plot particulier.
Ce système a été mis au point par
la société Cis-Bio International
en France). 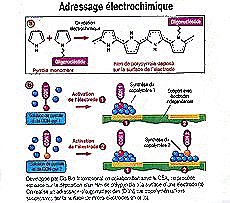
|
 Le
transfert d'oligonucléotide sur gel de polyacrylamide. Le
transfert d'oligonucléotide sur gel de polyacrylamide.
|
Les
sondes d'oligonucléotides sont transférées des plaques de
microtitration au gel grâce à un robot possédant une aiguille
de 1 nl dont le déplacement est dirigé par ordinateur.
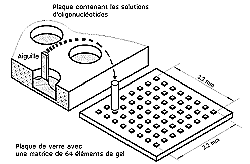
Figure
issue de Yershov et al.
|
 Le
transfert par LDI. Le
transfert par LDI.
|
La
LDI (Light Directed Immobilization) a été mise au point afin
d'immobiliser localement des oligonucléotides présynthétisés
et purifiés.
Deux méthodes appelées photolithographiques
ont été mise au point afon de fixer des oligomères modifiés
sur des surfaces de verre silanisées. 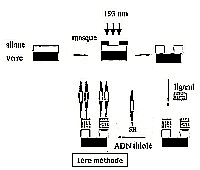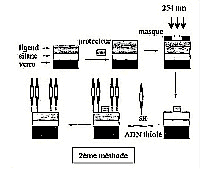
|
 L'Impression
de l'ADN. L'Impression
de l'ADN.
Une tête d'impression, comme celle d'une
imprimante, contient plusieurs solutions d'oligonucléotides, qu'elle
dépose ensuite très précisément sur le support. Cette technique,
développée par Incyte Pharmaceuticals
en collaboration avec Affymetrix,
permet aussi la synthèse in situ.
L'équipe de DeRisi et coll. (du laboratoire
de P.O. Brown) a mis au point cette technique dans laquelle les
sondes sont déposées au moyen d'un robot sur une lame de verre traitée
avec la poly-L-lysine.
3b.
Synthèse in situ.
La construction des sondes se fait par
dépôt de couches successives des quatre bases de l'ADN sur un support
en verre gràce à un système de masque.
|
Avec
ce procédé, utilisé par Affymetrix,
les sondes comportent au maximum 30 bases. En revanche, grâce
à une technique alliant des semi-conducteurs et la photolithographie,
la société Affymetrix
a réussi à synthétiser des milliers d'acides nucléiques sur
des supports. Cette technologie est basée sur la méthode VLSIPSTM;
(Very Large Scale Immobilised Polymer Synthesis), développée
par Fodor en 1991. 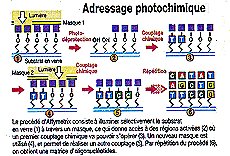
|
La
société Protogene, a mis au
point un système qui permet de déposer sur la grille de téflon de
la biopuce, les quatre bases désirées, grâce à des injecteurs piézo-électriques,
selon une technologie qui s'apparente à une synthèse d'ADN classique.
C'est l'équivalent d'une imprimante à jet d'encre où les quatre
couleurs sont en l'occurrence, les 4 bases : A, G, T, C.
Toutefois, le problème majeur de la synthèse
in situ reste l'incapacité à analyser et à corriger les éventuelles
erreurs introduites lors de la synthèse in situ sur le support.
|